30. November 2017
Virtuelle Realität für Bakterien
Ein interdisziplinäres Forscherteam hat einzelne Bakterien mit einem Computer verbunden, um einen biologisch-digitalen Hybridschaltkreis herzustellen – Ihre Studie wurde in Nature Communications veröffentlicht
© Remy Chait
Wissenschaftlern am Institute of Science and Technology Austria (IST Austria) ist es gelungen, das Verhalten einzelner Bakterien zu kontrollieren, indem sie diese an einen Computer koppelten. Mit Hilfe dieses Setups bauten sie einen genetischen Schaltkreis, der teils biologisch und teils digital ist. Das höchst interdisziplinäre Team umfasste die zwei gemeinsamen Erstautoren der Studie: den experimentellen Biologen Remy Chait und den Mathematiker Jakob Ruess, der mittlerweile am Institut Pasteur und am Inria Saclay in Frankreich tätig ist, sowie die IST Austria-Professoren Calin Guet und Gasper Tkacik. Ihr Experiment, bei dem sie die Genexpression in den Bakterien zum Oszillieren brachten und die Schwingungsmuster durch Anpassung der digitalen Kommunikation zwischen einzelnen Bakterien kontrollierten, diente als Machbarkeitsstudie. Eine mögliche Anwendung einer solchen biodigitalen Hybridtechnologie wäre das „Debuggen“ von komplexen biologischen Systemen auf eine ähnliche Weise, wie es bei komplexen Computercodes bereits geschieht: indem man die Komponenten einzeln testet, während man die Umgebung in einer Art virtueller Realität simuliert.
Wenn synthetische Biologen einen Mikroorganismus konstruieren wollen, der eine bestimmte Aufgabe erfüllt, der also zum Beispiel ein Krebsmedikament oder ein Antibiotikum als Teil seines Stoffwechselkreislauf herstellt, müssen Sie in der Regel eine erhebliche Anzahl von Änderungen am ursprünglichen Organismus vornehmen. Jede dieser Änderungen hat mehrere Auswirkungen, die mit den Auswirkungen aller anderen Änderungen wechselwirken und das Gesamtergebnis massiv verändern können. „Selbst wenn man versteht, was all die einzelnen Teile machen, weiß man nicht, was passiert, wenn man sie zusammenfügt“, erklärt Remy Chait die Herausforderung. „Es gibt Rückkopplungen zwischen ihnen, die den gesamten Schaltkreis unberechenbar machen.“
Eine mögliche Lösung für dieses Problem stammt aus der Softwareentwicklung und wird als Integrationstest bezeichnet. Bei diesem Ansatz wird jede Komponente einzeln getestet und ihre Wechselwirkung mit der Umgebung untersucht. Am besten simuliert man die Umgebung dabei durch einen virtuellen Raum und lässt die Komponente mit dieser virtuellen Welt interagieren. Genau diese Methode wollen die Forscher nun auch für biologische Systeme anwenden. „Biologische Systeme sind komplex und wir würden davon profitieren, wenn wir sie wie einen Computercode debuggen könnten,“ erklärt Remy Chait. „Beim Modul- und Integrationstest simuliert man die Umgebung und schließt die einzelnen Komponenten einzeln an, um sicherzustellen, dass sie wie vorgesehen funktionieren. Dann kombiniert man sie paarweise und beginnt von vorne. Auf diese Weise sieht man den Punkt, an dem die Rückkopplungen und Störungen das System zu stören beginnen, und kann es entsprechend anpassen“. Durch diese Methode kann der virtuelle Teil stetig reduziert werden, bis das System wieder voll biologisch ist – und dabei die gewünschte Funktion hat.
Die Forscher demonstrierten die Umsetzbarkeit von bio-digitalen Hybriden mit einem bio-digitalen Oszillator. In ihrem Aufbau produzieren modifizierte Escherichia coli-Zellen ein Protein, das blau-violett fluoresziert. Dieses farbige Licht bildet die Schnittstelle zur digitalen Komponente: Alle sechs Minuten misst der Computer, wie viel Licht die Zelle erzeugt, und akkumuliert proportional dazu ein virtuelles Signalmolekül. Wenn das Signal einen bestimmten Schwellenwert überschreitet, wird die Produktion des fluoreszierenden Proteins durch die Zelle abgeschaltet, und zwar mit Hilfe eines Projektors. Dieser beleuchtet die lichtempfindlichen Zellen mit rotem oder grünem Licht als „off“- oder „on“-Signal und verbindet somit die digitale Komponente wiederum mit den lebenden Teilen der Schaltung. „Die Zellen interagieren mit der simulierten Umgebung. Was sie tun, beeinflusst, was der Computer tut, und was der Computer tut, beeinflusst die Reaktion der Zelle. Wenn Sie Star Trek kennen, ist Ihnen das Holodeck sicher ein Begriff. Was wir gebaut haben, ist im Wesentlichen ein einfaches Holodeck für Gene von Mikroorganismen.“
Als die Forscher ihre hybriden Schaltkreise testeten, leuchtete die Zellpopulation in blau-violett, und dieses Leuchten oszillierte; allerdings mit Variationen zwischen den einzelnen Bakterien. Da die Forscher aber wollten, dass die Bakterien synchron schwingen, änderten sie die digitale Komponente entsprechend und bauten ein virtuelles Kommunikationsnetzwerk zwischen den Bakterien auf. Dieses verteilt einen Teil des virtuellen Signals zwischen benachbarten Zellen, und die Gruppe von Bakterien zeigte verschiedene Arten der gewünschten kollektiven Oszillation.
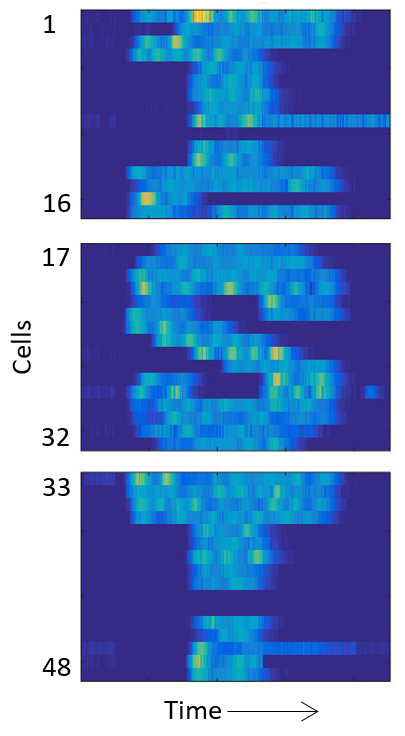
Eine andere Anwendung der Plattform ist die Rueckkopplungsregelung einzelner Zellen, welche die Genexpression des fluoreszierenden Gens so regelt, dass sie entlang einer vordefinierten Trajektorien verläuft. So könnten die Forscher eine Gruppe von Zellen dazu bringen, Bilder oder Buchstaben über die Zeit abzubilden (siehe Bild).
Publikation
R. Chait, J. Ruess, T. Bergmiller, G. Tkačik, C. C. Guet. 2017. Shaping bacterial population behavior through computer-interfaced control of individual cells. Nature Communications. DOI: /10.1038/s41467-017-01683-1



