14. September 2022
Proton Dominos als Startschuss für Leben
Universeller Mechanismus der molekularen Maschine Komplex I am ISTA erforscht
In einer neuen Publikation in Nature beschreiben ISTA-Professor Leonid Sazanov und sein Team einen universellen Mechanismus der Zellatmung, der in den meisten Spezies vorkommt, und kulminieren damit rund 20 Jahre an wissenschaftlicher Forschung. Diese entscheidenden Erkenntnisse auf dem Gebiet der Bioenergetik beruhen auf direkten Beobachtungen mittels kryogener Elektronenmikroskopie (Kryo-EM) am Institute of Science and Technology Austria (ISTA).
Die extra große molekulare Maschine namens Komplex I bringt die Energieversorgung in den Zellen in Gang. Zu verstehen, wie diese komplizierte Protonenpumpe funktioniert, ist für die Biologie von entscheidender Bedeutung. Aufgrund ihrer Rolle als Ausgangspunkt der Elektronentransportkette bei der Zellatmung wurde sie in den letzten Jahren eingehend untersucht. In einer neuen Publikation in Nature, die gestern Abend online veröffentlicht wurde, beschreiben Prof. Sazanov und sein Team den Mechanismus der Interaktion zwischen den verschiedenen Struktureinheiten von Komplex I: Um Energie für die Zelle zu gewinnen, beginnt hier der Elektronentransfer, und Protonen werden als Folge davon durch die Membran gepumpt, um eben der Zelle Energie zu liefern. Leonid Sazanov, ein prominenter Wissenschafter auf diesem Gebiet und einer der Autoren der Studie, leitet die Gruppe „Strukturbiologie von Membranproteinkomplexen“, die diese Studie am ISTA durchgeführt hat. Er fasst seine jüngsten Ergebnisse zusammen: „Wir haben jetzt eine detaillierte Erklärung für jeden Schritt des Mechanismus geliefert. Jede Protonenbewegung ist berücksichtigt. Das erklärt alle ungewöhnlichen Eigenschaften von Komplex I.“
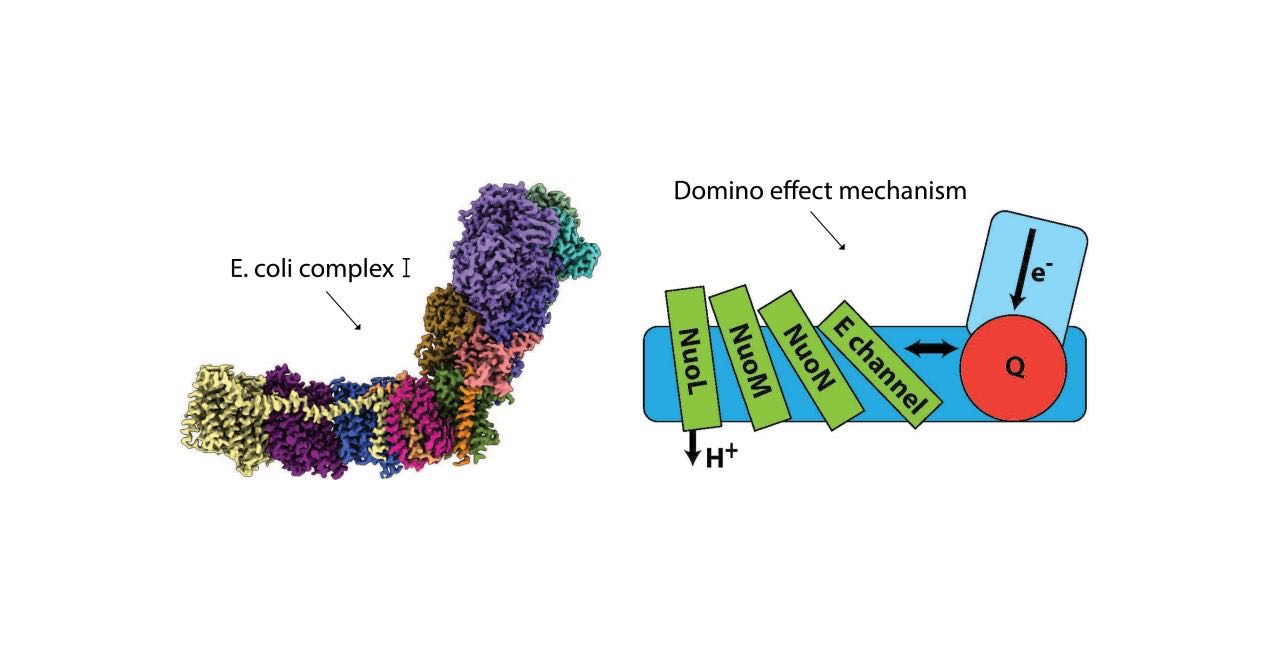
In ihrer Publikation schlagen Sazanov und sein Team einen detaillierten und dennoch robusten Mechanismus vor, der einen „Dominoeffekt“ von Protonentransfers und elektrostatischen Wechselwirkungen umfasst. Eine Vorwärtswelle (Stapeln der Dominosteine) setzt die Pumpe in Gang und die Rückwärtswelle (Fallen der Dominosteine) führt zum Ausstoß aller gepumpten Protonen aus einer distalen Untereinheit von Komplex I. Bisher war man davon ausgegangen, dass mehrere Untereinheiten Protonen ausstoßen. In dieser Studie wurde jedoch beobachtet, dass die meisten Protonen entlang der Membran und nicht über die Membran wandern, und zwar von einer Untereinheit zur nächsten und schließlich aus dem distalen Ende des Membranarms von Komplex I austreten.
Ein universeller Mechanismus
Die Sazanov Gruppe konzipierte ihre Studie so, dass die Ergebnisse frühere Erkenntnisse verifizieren und universelle mechanistische Prinzipien in den meisten Lebewesen – von Bakterien bis zu Säugetieren und auch in molekularen Maschinen, die dem Komplex I ähneln – aufzeigen. 2020 wurde eine Studie der Gruppe veröffentlicht, in der sie zum ersten Mal die atomare Struktur und die Mechanismen von Komplex I in den Mitochondrien von Säugetieren (Schafen) beschreiben. Komplex I in Säugetieren ist ein Megamolekül mit einem Gewicht von etwa 1 Megadalton (MDa), das seinerseits als Superkomplexe angeordnet ist. In der aktuellen Arbeit wurde E. coli, ein bakterielles Modell, verwendet, wo Komplex I etwa 0,5 MDa wiegt und nur konservierte Untereinheiten enthält.
Aus der Struktur von Komplex I ist bekannt, dass die Megastruktur ihre Form ändern kann: Sie kann ihre Arme entweder öffnen oder sie fest schließen. Im Säugetiermodell sah die Sazanov Gruppe sowohl offene als auch geschlossene Konformationen und legte deswegen nahe, dass Komplex I bei seiner Tätigkeit zwischen ihnen wechselt. Das warf viele Fragen auf, da unterschiedliche Konformationen bei anderen Arten nicht beobachtet wurden. Um alle Zweifel auszuräumen, so Sazanov, beschloss die Gruppe, als nächstes das Bakterium E. coli zu verwenden, da es evolutionär weit von den Säugetieren entfernt ist. Auch dieses Mal konnten sie in E. coli sowohl offene als auch geschlossene Zustände feststellen, ähnlich wie bei Säugetieren, jedoch mit einem entscheidenden Unterschied. Der geschlossene Zustand wurde nur während der Katalyse beobachtet, was zeigt, dass es sich tatsächlich um ein katalytisches Zwischenprodukt handelt. „Damit ist bewiesen, dass sowohl der offene als auch der geschlossene Zustand Zwischenzustände bei der Aktivität von Komplex I sind“, erklärt Sazanov.
Zugehöriges Video auf YouTube ansehen
„Es gibt Kernprotein-Untereinheiten, die von E. coli bis zu Säugetieren konserviert sind. Zuvor haben wir unseren vorläufigen Mechanismus im Säugetiersystem beschrieben, der aufgrund der vielen zusätzlichen Untereinheiten etwas kompliziert ist. Aber wir haben dasselbe in E. coli gesehen, was bedeutet, dass unser Mechanismus in allen Arten mit Komplex I universell ist“, so Sazanov.
Diese Arbeit bestätigt nicht nur die bahnbrechenden Erkenntnisse aus der Publikation von 2020, sondern schlägt auch einen universellen „Dominoeffekt“-Mechanismus der Protonenverschiebung vor, der letztlich unsere Zellen für Wachstum, Funktionen und Fortpflanzung befeuert. „Es handelt sich um einen universellen Kopplungsmechanismus von Komplex I und verwandten Enzymen“, so die Autor:innen in der Publikation, die am 22. September im Nature Journal erscheinen wird.
Neue Maßstäbe für Kryo-EM
Das hochmoderne kryogene Elektronenmikroskop am ISTA macht diese Erkenntnisse möglich. Mit seiner Hilfe kann die Sazanov Gruppe routinemäßig Beobachtungen mit einer Auflösung von bis zu 2 Ångström durchführen. Das reicht aus, um Wassermoleküle zu sehen und damit die Protonenübertragungswege zu identifizieren. Heute ist die Kryo-EM-Technologie ausgereift und schnell genug, um genügend Daten zu erhalten, um mechanistische Modelle von molekularen Maschinen zu entwickeln. Für jede Struktur wird nur etwa ein Monat benötigt, was mit Techniken wie der Röntgenkristallografie viel länger dauern würde.
Leonid Sazanov hat sich seit Beginn seiner wissenschaftlichen Laufbahn für große molekulare Maschinen interessiert. Als er in den späten 90er Jahren mit seiner Arbeit begann, war die Struktur des L-förmigen Komplex I weitgehend unbekannt. In der Folge arbeitete er viele Jahre lang mit Röntgenkristallographie, um zunächst die Struktur des hydrophilen Arms und dann die des Membranarms zu enträtseln, dessen genaue Funktionsweise er nun aufgedeckt hat. Sazanov merkt an, dass diese jüngste Arbeit auch mit Röntgenkristallographie umgesetzt werden könnte, was aber wahrscheinlich 20-30 Jahre länger dauern würde.
Der Erstautor der Studie und Postdoc in der Sazanov Gruppe, Vladyslav Kravchuk, führte die meisten Beobachtungen mit dem Kryo-EM durch – eine Aufgabe, die etwa drei Jahre dauerte. Es wurden mehr als 20 verschiedene Datensätze gesammelt, die notwendig waren, um Komplex I in verschiedenen Zuständen bei seiner Tätigkeit zu beobachten, zusammen mit Mutagenese Experimenten, die die Gruppe durchführte. Jeder Datensatz besteht aus fast 5.000 Mikrofotografien, die in der Regel innerhalb von 24 Stunden aufgenommen wurden. Die Mikrofotografien jedes Datensatzes liefern dann etwa eine Million Einzelpartikelbilder, die ausgewählt und mit Hilfe eines Clusters von ISTA-Hochleistungscomputer rechnerisch analysiert werden. „Eine der größten Herausforderungen des Projekts war die sorgfältige Analyse der Kryo-EM-Daten, da Komplex I recht flexibel ist und verschiedene Konformationszustände annimmt. Nur durch die umfangreiche, sorgfältige und manchmal unkonventionelle Analyse gelang es uns, den oben erwähnten geschlossenen Zustand vollständig aufzuklären. Dies klärte viele Fragen und Debatten in diesem Bereich und wird weiterhin als Maßstab für die Kryo-EM-Datenanalyse von Komplex I und verwandten Molekülen dienen“, sagt Vladyslav Kravchuk.
Publikation:
https://www.nature.com/articles/s41586-022-05199-7
Kravchuk, V. et al. 2022. A universal coupling mechanism of respiratory complex I. Nature.
DOI: 10.1038/s41586-022-05199-7
Projektförderung:
Dieses Projekt wurde von den Scientific Service Units (SSU) des ISTA durch Ressourcen der Electron Microscopy Facility (EMF), der Life Science Facility (LSF) und des ISTA-Hochleistungscomputer-Clusters unterstützt. Der erste Autor war Empfänger eines DOC-Stipendiums der Österreichischen Akademie der Wissenschaften am Institute of Science and Technology, Austria. Zwei der Autoren werden durch den ERC Advanced Grant für Professor Leonid Sazanov finanziert.



