24. September 2020
Rätsel um riesige Protonenpumpe gelöst
Mechanismus, wie Komplex I der Atmungskette Proton pumpt, endlich verstanden – Studie in Science erschienen
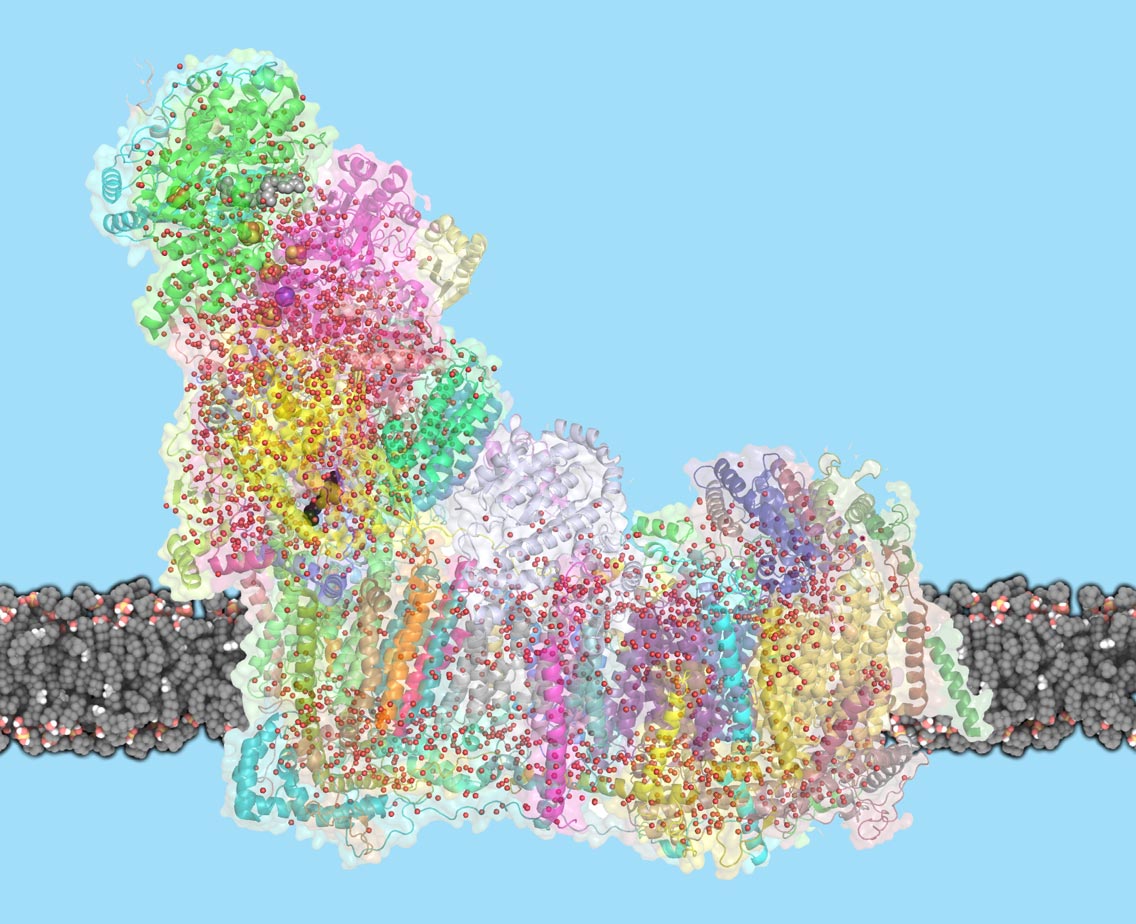
Mitochondrien sind die Kraftwerke unserer Zellen, sie erzeugen Energie, die das Leben unterstützt. Eine riesige molekulare Protonenpumpe, genannt Komplex I, ist dabei entscheidend: Sie setzt eine Kette von Reaktionen in Gang und erzeugt einen Protonengradienten, der die Erzeugung von ATP antreibt, dem Brennstoff der Zelle. Trotz der zentralen Rolle von Komplex I ist der Mechanismus, mit dem Komplex I Protonen über die Membran transportiert, bisher unbekannt. Nun haben Leonid Sazanov und seine Gruppe am Institute of Science and Technology Austria (IST Austria) das Rätsel gelöst, wie Komplex I funktioniert: Änderungen in der Konformation kombiniert mit elektrostatischen Wellen bewegt Protonen in die mitochondriale Matrix. Das ist das Ergebnis einer heute im Fachjournal Science veröffentlichten Studie.
Komplex I ist das erste Enzym in der Atmungskette, einer Reihe von Proteinkomplexen in der inneren Mitochondrienmembran. Die Atmungskette ist für den größten Teil der Energieproduktion der Zelle verantwortlich. In dieser Kette bauen drei Membranproteine einen Protonengradienten auf; sie bewegen Protonen vom Zytoplasma der Zelle in den mitochondrialen Innenraum, die so genannte Matrix. Die Energie für diesen Prozess stammt hauptsächlich aus dem Elektronentransfer zwischen NADH-Molekülen, die aus unserer Nahrung stammen, und Sauerstoff, den wir einatmen. Die ATP-Synthase, das letzte Protein in der Kette, nutzt diesen Protonengradienten, um ATP zu erzeugen. Komplex I ist nicht nur wegen seiner zentralen Rolle im Leben bemerkenswert, sondern auch wegen seiner schieren Größe: Mit einem Molekulargewicht von 1 Megadalton ist der eukaryotische Komplex I eines der größten Membranproteine. Seine Größe macht es auch schwierig, Komplex I zu untersuchen. Sazanov und seine Gruppe waren 2016 die ersten, die die Struktur des Komplex I bei Säugetieren lösten, nachdem sie 2013 die Struktur eines einfacheren bakteriellen Enzyms aufgeklärt hatten. Doch der Mechanismus, mit dem Komplex I Protonen über die Membran bewegt, ist nach wie vor umstritten. „Eine Idee war, dass ein Teil von Komplex I wie ein Kolben funktioniert, der Kanäle öffnet und schließt, durch die dann Protonen wandern”, erklärt Sazanov. „Eine andere Idee war, dass Moleküle im Zentrum von Komplex I als Antrieb wirken. Es stellt sich heraus, dass ein noch ungewöhnlicherer Mechanismus am Werk ist”.
Wasserader hilft Protonen, die Membran zu kreuzen
In der Vergangenheit haben Sazanov und seine Gruppe gezeigt, dass der L-förmige Komplex I aus hydrophilen und Membranarmen besteht. Im hydrophilen Arm tunneln Elektronen von NADH zu Chinon, dem hydrophoben Elektronenträger. Der Membranarm, in dem die Protonentranslokation stattfindet, besteht aus drei ähnlichen Untereinheiten mit Strukturen, die mit Antiportern verwandt sind, sowie einer Untereinheit, die einen Chinon-bindenden Hohlraum enthält. In diesem Hohlraum überträgt Komplex I zwei Elektronen pro katalytischem Zyklus auf Chinon, das die Elektronen weiter an die Komplexe III und IV abgibt. Es war jedoch ein Rätsel, wie die Wechselwirkung zwischen Elektronen und Chinon vier Protonen pro Zyklus über die Membran bewegen kann, da die antiporterartigen Untereinheiten weit vom Chinon-Hohlraum entfernt sind. Um dieses Rätsel zu lösen, führten Sazanov und sein Team Kryo-EM am Komplex I von Schafen durch. In einer Kraftanstrengung löste der Doktorand Domen Kampjut 23 verschiedene Strukturen des Komplexes I, die unter unterschiedlichen Bedingungen erhalten wurden. Durch Hinzufügen von NADH und Chinon konnten die Forscher Bilder von Komplex I bei der Arbeit aufnehmen, während es die Konformation zwischen den beiden Hauptzuständen veränderte. Dank der hohen Auflösung konnten sie die Wassermoleküle im Inneren des Proteins auflösen, die für den Protonentransfer unerlässlich sind. Sie fanden heraus, dass viele Wassermoleküle in der Mittelachse des Membranarms eine Möglichkeit für Protonen bieten, zwischen polaren Rückständen und Wassermolekülen zu hüpfen, und so Bahnen entlang und durch die Membran bilden.
Aber nur in einer Untereinheit, die am weitesten von Chinon entfernt ist, hüpfen Protonen tatsächlich so über die Membran. Die beiden anderen Untereinheiten bieten eine Kopplung zwischen der am weitesten entfernten Untereinheit und Chinon. Wenn der Bindungshohlraum auf Chinon „wartet”, blockiert eine Helix die Wasserader in der Mittelachse. Wenn Chinon in der Bindungshöhle bindet, ändert sich die Protein-Konformation um diesen Bereich dramatisch und die Helix rotiert. Nun verbindet die Wasserader alle Membran-Untereinheiten des Komplexes I, und zwei Protonen werden dem Chinon zugeführt, um dessen Reduktion zu vervollständigen. Dieser Schlüsselteil des Mechanismus erzeugt eine Ladung in der Nähe des ersten Antiporters und setzt eine elektrostatische Welle von Wechselwirkungen zwischen geladenen Resten in Gang, die sich entlang der Antiporter ausbreitet und zur Translokation von insgesamt vier Protonen führt. „Wir zeigen, dass im Komplex I ein neuer und unerwarteter Mechanismus am Werk ist. Eine Mischung aus Konformationsänderungen und einer elektrostatischen Welle pumpt Protonen über die Membran”, erklärt Sazanov. „Dieser Mechanismus ist höchst ungewöhnlich, da er die Rotation einer ganzen Helix im Inneren des Proteins beinhaltet. Es scheint ein bisschen übertrieben, aber wahrscheinlich trägt das dazu bei, dass der Mechanismus robust ist.“
Die neue Forschung ergänzt die in den letzten zwei Monaten veröffentlichten Studien der Sazanov-Gruppe über den Mechanismus des Protonenpumpens im Bakterienkomplex I (Nature Communications) und über die Struktur der MRP-Antiporter, aus denen sich der Komplex I entwickelt hat (eLife).



